Eine sinnvolle Dateistruktur für R-Projekte
Haben Sie schon mal versucht, sich in der Dateistruktur eines bereits bestehenden Projekts zurechtzufinden, in einem "historisch gewachsenen" Verzeichnis relevante von veralteten Dateien zu unterscheiden, oder herauszufinden, in welcher Reihenfolge vorhandene Skripte ausgeführt werden müssen?
Damit dies leichter wird, ist eine konsistente Datei- und Ordnerstruktur in Ihren Projekten sehr hilfreich. Dieser Artikel richtet sich an Leserinnen und Leser, die erste Erfahrungen mit R, RStudio und eventuell mit der Entwicklung von R-Paketen mitbringen. Vor allem, wenn Sie R im Team nutzen, ist dieser Artikel für Sie interessant.
Ähnlich wie beim Coding-Style ist jedoch vor allem wichtig, dass sich alle Team-Mitglieder auf gemeinsame Regeln einigen - und nicht, wie diese Regeln genau aussehen.
Eine nützliche Dateistruktur
Es gibt natürlich nicht die eine oder die beste Dateistruktur. Die folgende Abbildung zeigt unsere Lösung für die Dateistruktur, die sich in unseren Data Science-Projekten bewährt hat:
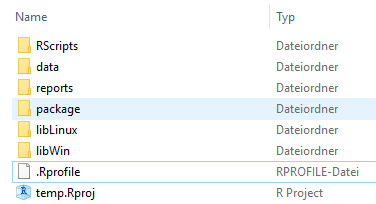 Abb. 1 - Eine nützliche Dateistruktur
Abb. 1 - Eine nützliche Dateistruktur
Wenn Sie mit RStudio arbeiten, wird Ihnen die .Rproj-Datei vielleicht bekannt vorkommen. Diese Datei bestimmt das Projekt- und Arbeitsverzeichnis für R-Sitzungen. Dies hat den Vorteil, dass alle Dateipfade relativ zum Arbeitsverzeichnis des Projekts gesetzt werden können. Dadurch können Sie den gesamten Projektordner verschieben und die Pfade in Ihrem Code funktionieren trotzdem noch.
Der erste Ordner heißt RScripts. Er enthält – wie der Name vermuten lässt - alle R-Skripte, z.B. zur Datenaufbereitung und Datenanalyse. Wir empfehlen, die Skripte der Reihenfolge nach zu nummerieren, in der sie ausgeführt werden müssen. Zum Thema Skripte sehen Sie sich auch gern den Artikel INWT-Guidelines für R-Code an.
Skripte erzeugen häufig Output, z.B. Daten in tabellarischer Form, Grafiken, .tex-Dateien usw. Diese Outputs werden dann entweder in einem Ordner data oder results gespeichert. Die Unterscheidung dieser beiden Ordner ist manchmal nicht ganz eindeutig, aber generell enthält data alle Daten, auf denen die Datenanalyse basiert. Der Ordner results dient zum Speichern der aus den Skripten resultierenden Endergebnisse, aber nicht der Zwischenschritte. Dieser Ordner befindet sich entweder im Ordner data oder direkt im Projektverzeichnis.
Dateien im Ordner results werden häufig zum Zwischenspeichern von Ergebnissen genutzt, die später in einem R Markdown-Report benötigt werden. Obwohl knitr ein eigenes Caching-System enthält, ist es in vielen Projekten besser, einzelne Berechnungen zu entkoppeln, damit sich Reports schnell und unkompliziert kompilieren lassen.
Alle Daten (Rohdaten und aufbereitete Daten) befinden sich im Ordner data. Dazu gehören verschiedene Dateiformate wie .Rdata, .xlsx oder .csv. Diese Daten werden vom R-Paket getrennt gespeichert, damit man Änderungen am Paket schneller vornehmen kann. Dies ist besonders relevant, wenn die Dateien sehr groß sind. Sie werden auch nicht von der Versionskontrolle erfasst. Meistens sind diese Dateien Outputs von R-Skripten, die zeitaufwändige Datentransformationen durchführen oder Daten aus externen Ressourcen extrahieren (z.B. Datenbanken oder Crawler). Daher können diese Dateien immer mit Hilfe von Skripten rekonstruiert werden. Dieser Ordner wird nie als permanenter Dateispeicher verwendet.
Der Ordner reports enthält die Reports, die wir mit R Markdown erstellen. Dafür haben wir übrigens unsere eigenen R Markdown-Vorlagen und ein ggplot2-Theme, sodass die Reports direkt unserem Corporate Design entsprechen.
Wo liegt das Paket?
Zu unseren Projekten gibt es quasi immer ein eigenes R-Paket. R-Pakete haben ihre eigene spezifische Datei- und Ordnerstruktur, aber wo im Projekt sollten diese Dateien liegen? Grundsätzlich gibt es zwei Möglichkeiten: Entweder werden alle Paketdateien in einem eigenen Ordner gespeichert (zum Beispiel package wie in Abb. 1), oder direkt im Projektverzeichnis (wie in Abb. 2).
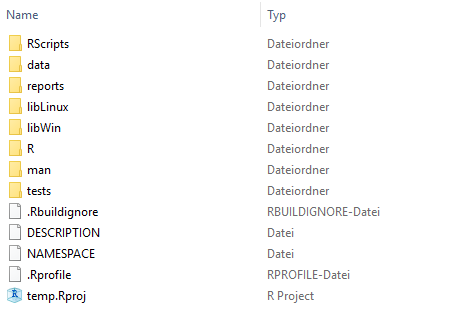 Abb. 2 - Eine nützliche Dateistruktur mit R Paket
Abb. 2 - Eine nützliche Dateistruktur mit R Paket
Welche Variante praktischer ist, hängt von der Art des Projekts ab. Auch eine reine Datenanalyse wird immer von einem Paket unterstützt, denn selbst wenn die Funktionen in keinem anderen Projekt wiederverwendet werden, will unser Code sauber dokumentiert und getestet werden. Außerdem müssen Paketabhängigkeiten im Team und auf den einzelnen Computern installiert und gepflegt werden. In einem reinen Datenanalyse-Projekt, z. B. der Vorhersage von Verkaufszahlen oder der Berechnung eines Customer Lifetime Value, befindet sich das Paket in seinem eigenen Ordner. Dadurch wird auch ein Konflikt vermieden: Normalerweise brauchen Sie in einem Analyseprojekt einen Ordner data. Pakete enthalten aber auch einen Ordner namens data, sodass R beim Erstellen des Pakets versuchen würde, Ihren Datenordner zu berücksichtigen, was Sie vermutlich nicht wollen.
In Projekten, in denen die Paketentwicklung die Hauptaufgabe ist, befindet sich das Paket direkt im Projektverzeichnis. Dies immer dann der Fall, wenn Pakete Tools bereitstellen, die projektübergreifend verwendet werden – z.B. unser INWTUtils-Paket.
Paketentwicklung: Arbeiten in einer Testumgebung
Wir installieren alle R-Pakete - unsere eigenen und die von CRAN - auf einem Netzlaufwerk. So greift das ganze Team auf die gleichen Paketversionen zu. Aber wenn jemand an einem unserer eigenen Pakete arbeitet, soll natürlich nicht jede kleine Änderung sofort auf dem Netzlaufwerk erscheinen: Sie könnte noch Fehler enthalten und die Arbeit der Kollegen stören.
Um dieses Problem zu lösen, arbeiten wir beim Weiterentwickeln unserer Pakete in einer Testumgebung bzw. einer sog. Sandbox („Sandkasten“): Wenn wir das Paket neu bauen, wird es nicht auf das Netzlaufwerk geschrieben, sondern in einen bestimmten lokalen Ordner, den Sie in Abbildung 1 sehen: libLinux bzw. libWin. Auf diese Weise bleibt alles, was wir ausprobieren, in einer abgeschlossenen Umgebung. Erst wenn alle Änderungen abgeschlossen und getestet sind, wird die aktualisierte Paketversion auf dem Netzlaufwerk installiert.
Aber woher weiß RStudio nun, wohin das Paket installiert werden soll, wenn wir es bauen? Das ist in der .Rprofile-Datei hinterlegt. Diese wird immer ausgeführt, wenn Sie R öffnen. Unsere .Rprofile ändert das Standardinstallationsverzeichnis in Ihrem R-Projekt zu libLinux bzw. libWin. Eine Beispieldatei können Sie sich hier ansehen. Die Ordner libLinux und libWin sind übrigens auch gut geeignet, um Pakete zu installieren, die Sie einfach mal ausprobieren möchten, ohne das Netzwerklaufwerk „vollzumüllen“.
Doch auch wenn Sie nicht auf ein geteiltes Paketverzeichnis auf einem Netzlaufwerk zugreifen, kann das Arbeiten in einer Testumgebung Vorteile bringen: Sie können zum Beispiel an einem eigenen Paket arbeiten, ohne die stabile Version in Ihrer Benutzerbibliothek zu beeinflussen.
Fangen Sie einfach an!
Es ist und bleibt oft schwierig, sich in einem Projekt zu zurechtzufinden, in dem viele Menschen ihre Spuren hinterlassen haben. Aber es wird einfacher, wenn die Dateistruktur einheitlich und durchdacht ist. Und das zwingt Sie auch, so zu arbeiten, dass Ihre Kollegen sich nachher noch in Ihren Dateien und Ordnern zurechtfinden.
Zu Beginn eines Projekts verwenden wir das Paket INWTUtils, um die in diesem Artikel beschriebene Dateistruktur zu erstellen. Auf diese Weise kann eine einheitliche Struktur ganz einfach im gesamten Team umgesetzt werden.
